运用的文件是4个,myCV是群体结构文件,myKI是亲缘关系文件,myY是表型文件,myG是基因型文件。
根据之前测试,只加myG和myY文件可以运行,加入群体结构和亲缘关系文件出现错误,于是修改群体结构和亲缘关系文件。
首先,建立一个空的myCV文件,如下:

出现以下报错,该进度在51%。
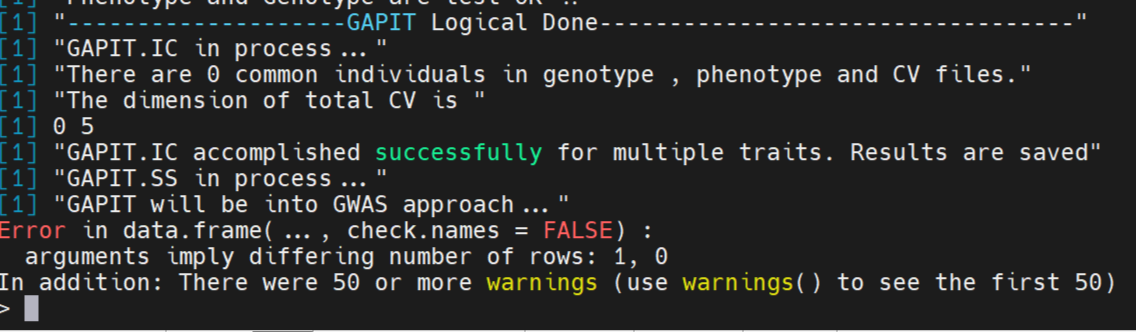
于是,修改了CV文件,创建一个260行4列的数据框。
#创建一个数据框
new_cv <- data.frame(matrix(nrow = 260, ncol = 4))
#填充数据
mydata <- read.table("Rp_CV_3.txt", header = FALSE, sep = "\t")
new_cv <- mydata
write.table(new_cv, file = "new_CV_file.txt", sep = "\t", row.names = FALSE, col.names = FALSE)使用新的CV文件,运行程序,出现新的报错,该报错在进度53%
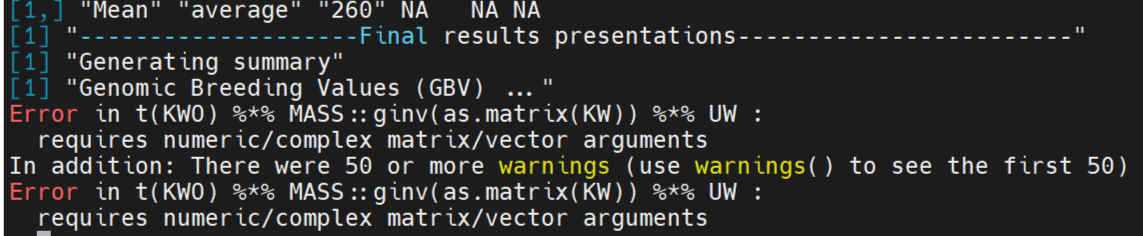
报错表明:不是一个矩阵
针对KINSHIP文件进行修改
首先,使用以下命令:
myKI <- read.table("KINSHIP.txt", header = FALSE)
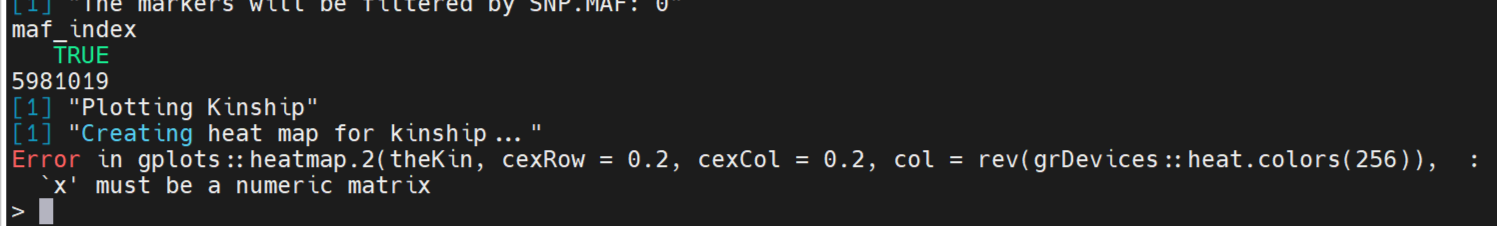
myKI_matrix <- as.matrix(myKI)出现报错,该进度在53%之前
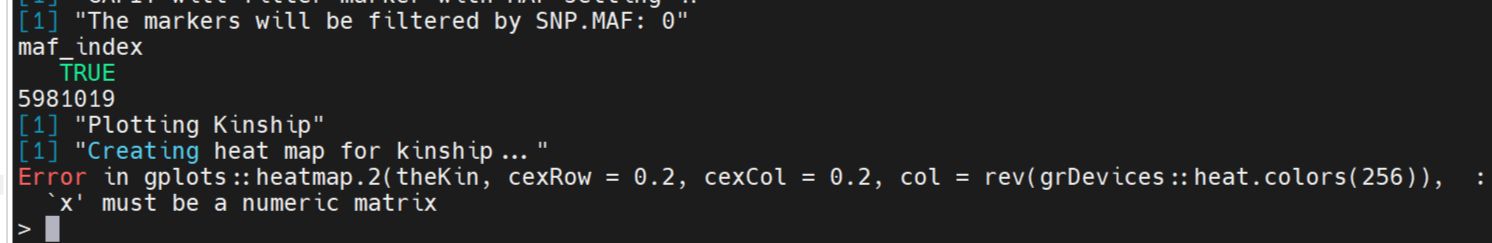
这个报错信息来自 gplots::heatmap.2 函数,并且它告诉你 'x' must be a numeric matrix,意思是说你传递给 heatmap.2 函数的 x 参数必须是一个数值型矩阵。
于是,创建一个260行,261列的矩阵
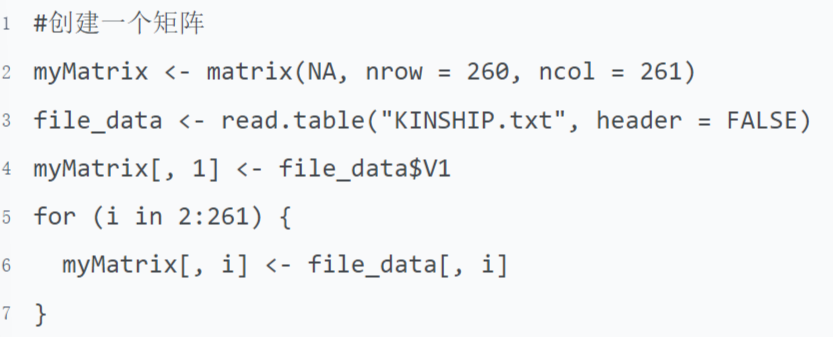
结果,出现上述一样的报错。
猜测,可能kinship文件还是存在问题,于是检查自己的文件类型,对比示例数据和自己的数据。
#查看示例文件如下:
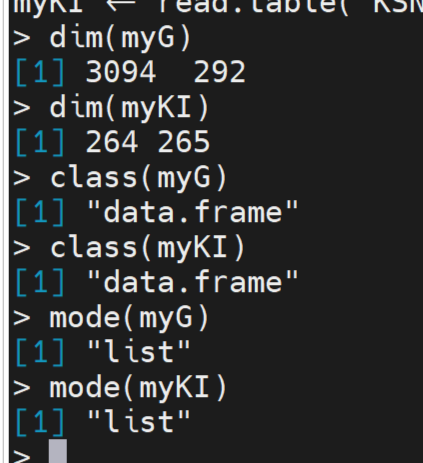
#查看自己的文件如下:
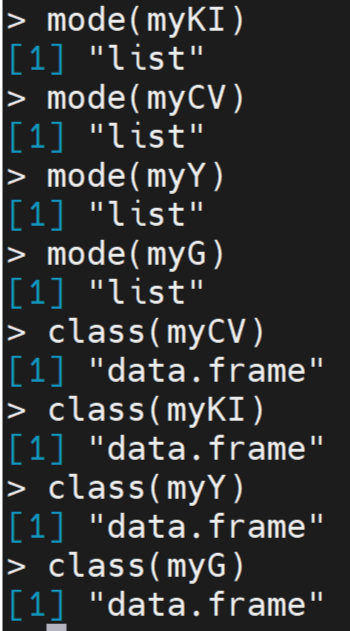
发现自己的文件和示例文件一致。然后,检查文件的内容,发现KINSHIP文件中存在空格,检查文件中的空格是否符合要求。
#输入命令检查文件
cat -T KINSHIP.txt
#如果文件中是tab键的空格,则会输出^I,发现KINSHIP文件中不是TAB键空格,于是转换文件中的内容。
#两个方法,方法一:
sed 's/ / /g' KINSHIP.txt > KINSHIP.new.txt
cat -T KINSHIP.new.txt#还是没有显示^I
#方法二:
tr ' ' '\t' < KINSHIP.txt > KINSHIP.new_2.txt
cat -T KINSHIP.new_2.txt #这个方法成功!#解决KINSHIP文件后,等待测试。