参考:Bulik-Sullivan, B., Loh, PR., Finucane, H. et al. LD Score regression distinguishes confounding from polygenicity in genome-wide association studies. Nat Genet .47, 291–295 (2015). https://doi.org/10.1038/ng.3211
这款工具主要是区分飘逸的QQ图正不正常。
多基因性(即若干较小的基因效应)和干扰因素引起的偏差(如隐性关联 ,群体分层等)都会造成检验的统计量的分布偏高。于是,开发了LD Score (LD分数回归),检查测量统计量与连锁不平衡之间的关系来量化每个不分的贡献。
LDSC本质是一个线性回归,其输入数据为GWAS的分析结果,回归的自变量为SNP位点的LD score值,因变量是该算法的核心,自定义的一个符合卡方分布的统计量,通过线性回归拟合LD score和卡方统计量的关系,从而判断GWAS分析结果中是否存在混淆因素。
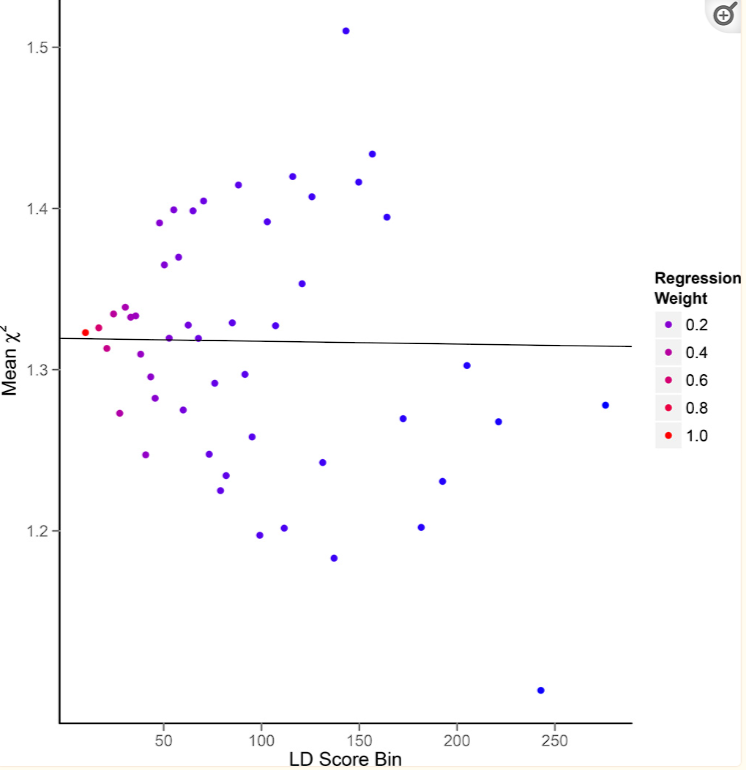
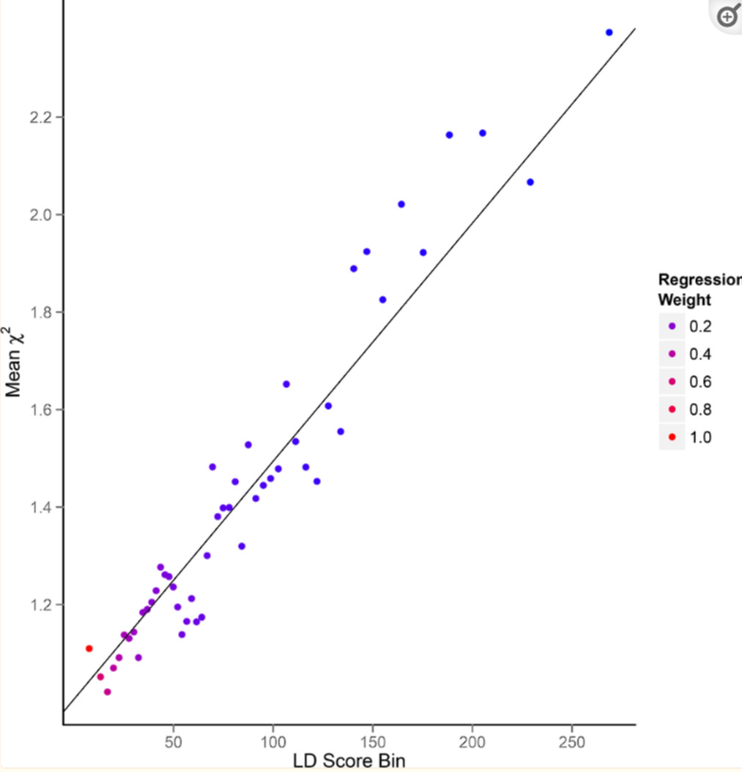
主要通过LDSC工具计算基因组数据的LD回归截距,如果是基因多效性,那么截距会接近1(比如1.004),如果是群体分层等混淆因素引起的,那么LD回归截距就会远离1(比如1.30)。
(a)具有混杂因素的QQ图
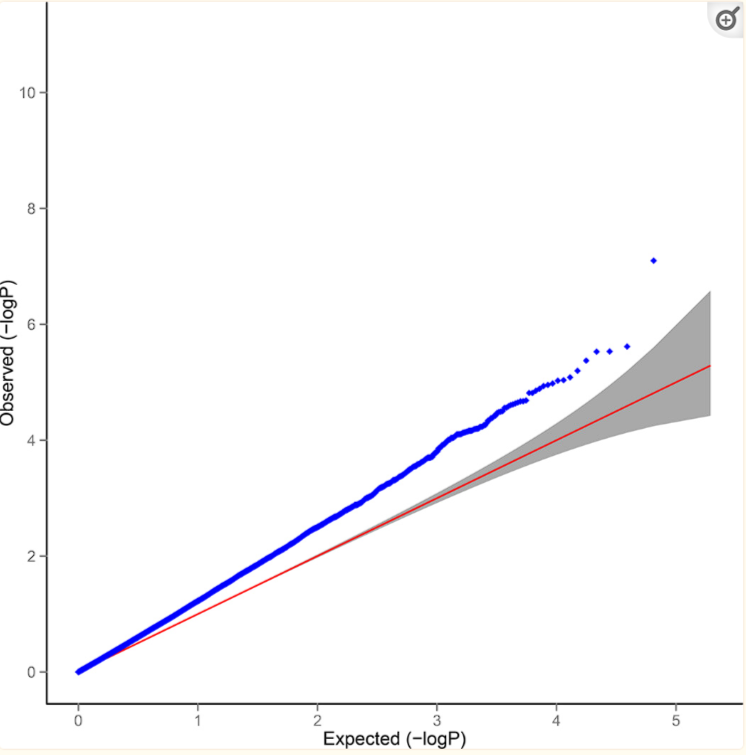
(b)具有混杂因素的LD截距图
(c)具有基因多效性的QQ图
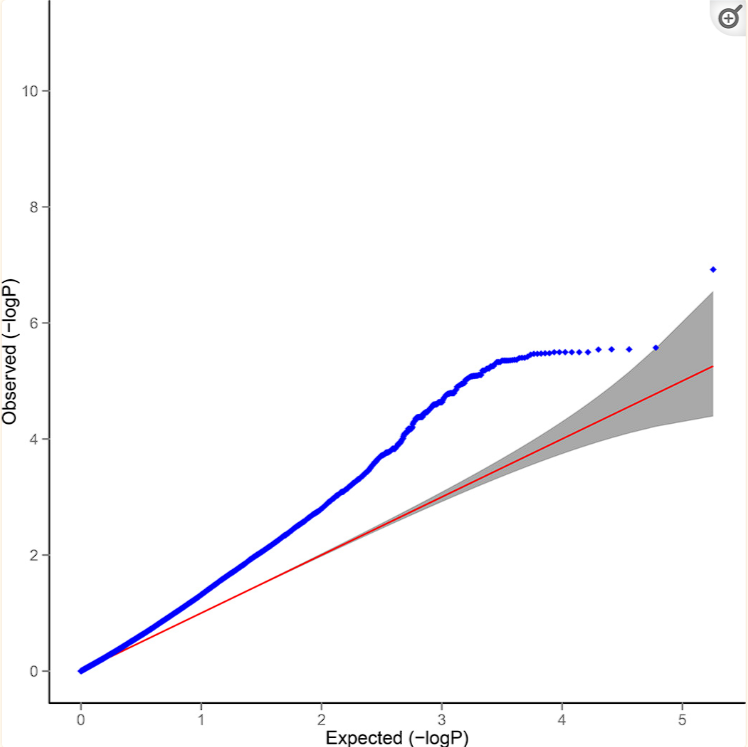
(d)具有基因多效性的LD截距图
总结:截距很接近1,就不用管QQ图好不好看了;远离1说明基因型可能有问题,检查一下PCA、群体分层有没有控制好、是否混了很多有亲缘关系的样本在里面。
认为可以作为GWAS结果的一个验证。
下载以及安装:这个工具主要以python为脚本,配置conda环境。