Description
侃大山,随便聊聊。有啥新奇特的Idea都可以分享。
侃大山,随便聊聊。有啥新奇特的Idea都可以分享。
在网站上上传文件的时候,会报错.
【金山文档 | WPS云文档】 2024_12_30日志(香榧性别逆转猜想)
需要实现在新建的一个视图中让用户按照特定的字段实现内容搜索功能。
- 在视图的编辑页面(Structure > Views > Edit)中,Filter criteria 表示对该类型的内容进行筛选,并显示到表格中。
- 如果需要对特定字段进行:
- 首先需要再在Filter criteria中添加该字段。
- 其次,为了将筛选条件暴露给用户时,则需要对字段进行设置。即在字段的编辑页面,勾选 Expose this filter to visitors, to allow them to change it 。
- 保存,会在视图中显示筛选条件。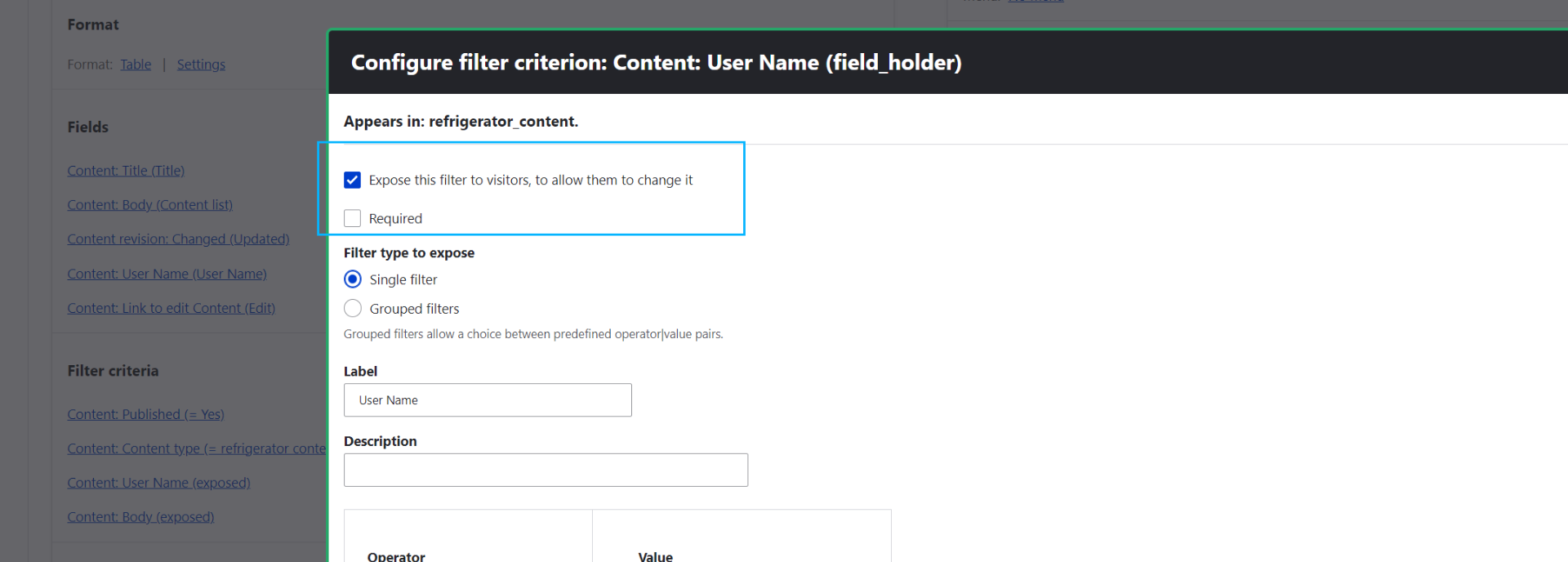
转录组的数据、删除的id名称统计、替换的id名称
1、在/home/replace_geneid目录下,先根据鹏哥给的abandoned.gene.id.txt文件,剔除转录组中所有已经被删掉的id。python代码为split_genes.py,输入命令python split_genes.py。生成included_genes文件和excluded_genes文件。